Субекти
Обобщение
Лошо проученият пикорнавирус, човешки пареховирус 3 (HPeV3) причинява неонатален сепсис без налични терапии. Нашата собствена структура с резолюция 4.3-Å на HPeV3 и резолюция 15 Å в комплекси с човешки моноклонални антитела Fab демонстрира очакваната структура на пикорнавирусния капсид с три различни характеристики. Първо, 25% от генома на HPeV3 РНК на 60 места е силно подреден, както е потвърдено от асиметрична реконструкция, и взаимодейства със запазени региони на VP1 и VP3 капсидните протеини. Второ, терминът VP0 N стабилизира вътрешната повърхност на капсида, за разлика от други пикорнавируси, при които при изхвърляне като VP4, той образува канал за транслокация на РНК. И накрая, хидрофобният джоб на VP1, мястото на свързване на антипикорнавирусното лекарство, плеконарил, е блокиран и следователно неподходящ за антивирусно развитие. Взети заедно, тези резултати предполагат посока за развитието на неутрализиращи антитела, антивирусни лекарства, базирани на взаимодействия РНК-протеин, и дисекция на вирусно събрание въз основа на РНК нуклеация.
Въведение
Picornaviridae е семейство от малки, позитивно усетени, икозаедрични симулации, едноверижни РНК вируси. Пареховирус А е вид в това семейство с 16 генотипа и се свързва главно с леки инфекции при хората, особено при децата. Новият патоген, човешки пареховирус 3 (HPeV3), обаче, може да причини сериозни инфекции в централната нервна система, като менингит 1, и е водеща причина за неонатален сепсис 2. Няма налични антивирусни средства или ваксини за борба с HPeV инфекцията. За разлика от много други пикорнавируси, HPeVs са слабо характеризирани както по отношение на структурата, така и по отношение на функцията, с изключение на HPeV1, където рецепторът 3 е известен. Големите разлики в тропизма, показани от HPeV3 в сравнение с другия HPeV, правят изключително важно изследването на структурните свойства на HPeV3 за по-добро разбиране на неговата патогенеза и възможното свързване с рецепторите.
Използвахме криоелектронна микроскопия и реконструкция на изображението, за да анализираме структурата на HPeV3 самостоятелно и в комплекс с човешко моноклонално антитяло Fab. Структурата на вирион показва, че VP1 джобно свързващи лекарства, като плеконарил, е малко вероятно да се свържат с HPeV; че VP0 е важен протеин за стабилизиране на вътрешната повърхност на капсида и накрая, че HPeV сборът вероятно се контролира от множество взаимодействия на генома с капсида, чрез консервирани аминокиселини във VP1 и VP3 и структури на стволови и контурни в РНК. Ние изолираме и охарактеризираме човешко моноклонално антитяло, специфично за HPeV3, което може да бъде много полезно за подобряване на диагностиката на вируси и изучаване на взаимодействията между вируса и гостоприемника.
Резултати и дискусия
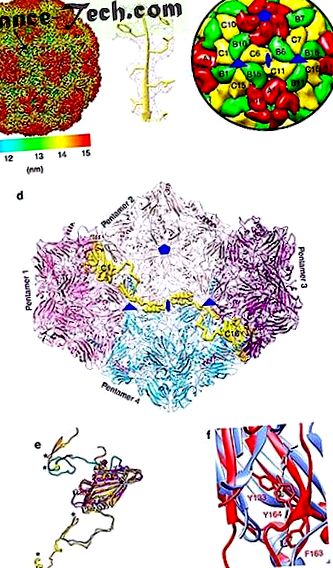
Структура на HPeV3
Изображение в пълен размер
Подредена структура на HPeV3 РНК
Изображение в пълен размер
Изображение в пълен размер
Хидрофобен джоб VP1
Антивирусното лекарство, плеконарил и неговите производни обикновено блокират ентеровирусни инфекции като EV-D68 (справка 4). Тези лекарства изхвърлят липида ("джобен фактор"), присъстващ в хидрофобния джоб на VP1 β-цевта и блокират РНК покритието чрез стабилизиране на капсида 4. Каналът към хидрофобния джоб в HPeV3 VP1 е блокиран от три големи запазени странични вериги Y133, F163 и Y164 (фиг. 1е и 3) в сравнение с EV-D68 (реф. 4). Следователно можем да обясним наблюденията, че плеконарилът не работи в клиника 2 и да прогнозираме, че подобни имитатори на джобни фактори няма да работят срещу нито един от секвенираните досега HPeV.
HPeV3 - Fab сложна структура
Изолирахме специфично за HPeV3 човешко моноклонално антитяло, AT12-015, което се свързва с HPeV3 изолати 152037, A308/99 и два клинични изолата в заразените клетки. Той не неутрализира 152037 в анализ, базиран на клетъчни линии Vero. Структурата на HPeV3 изолат 152037 беше разрешена в комплекс с Fab AT12-015 (допълнителна таблица 1; фиг. 4а; допълнителна фигура 1). Fab молекулите разпознават конформационен епитоп по ръба на цевта (фиг. 4а). Приносът идва от региони както във VP3 (hZβB, βChA, βEβF и βGβH контури), така и във VP1 (βBβC, βCβD и βEβF, C), запазени главно в HPeV3, което обяснява специфичността на антитялото към този генотип (фиг. 3) . Fab пръстовият отпечатък обхваща терминала VP1 C, където много други HPeVs съдържат RGD мотив, за да се свържат с техните интегринови рецептори (Фиг. 3 и 4b) 3. Тъй като AT12-015 е специфичен за HPeV3 и не се свързва с нито един от другите тествани генотипове на HPeV, той може да бъде много полезен при напредването на диагностиката на вирусите и изучаването на взаимодействията между вируса и гостоприемника. Допълнително сравнение с епитопите на неутрализиращите пациентски серуми може да ни помогне да разберем механизма на неутрализация при пациенти 13 .
( да се ) Радиално цветно изоповърхностно представяне на комплекса HPeV3-Fab AT12-015 при разделителна способност 15 Å, показана при 1,5σ над средния праг. Fab молекули (червени) се свързват около района на каньона. ( б ) HPeV3 пътна карта. Отпечатъкът на Fab (червен контур) се преобразува в VP3 (зелен) и VP1 (червен).
Изображение в пълен размер
Като цяло тази работа показва множество стабилизиращи РНК-капсидни взаимодействия като нова цел за антипареховирусни лекарства. Ние разкриваме необичайната стабилност на тези капсиди на ниво интра-пентамер чрез VP3 взаимодействия и на интер-пентамера от VP0 и предполагаме, че механизмът на HPeV покритие на генома ще се различава значително от другите пикорнавируси.
Методи
Пречистване и инактивиране на вируси.
HPeV1-Harris и HPeV2-2008 бяха предоставени от Холандския национален институт за обществено здраве и околна среда, Билтовен, Холандия. HPeV4-251176, HPeV5-552322, HPeV6-550389 и два HPeV3 клинични изолата са получени от Лабораторията по клинична вирусология, Академичен медицински център, Амстердам, Холандия. За вирусни запаси HPeV1, HPeV2, HPeV3 A308/99 (тип подарък Hiroyuki Shimizu и Miyabe Ito) и HPeV4-6 бяха култивирани в клетъчна линия HT29 в минималната основна среда на Eagle с L-глутаминова киселина (0, 2 ×), несъществена Аминокиселини (1x), стрептомицин (0.1 μg ml -1), пеницилин (0.1 μg ml -1) и 2% топлинно инактивиран FBS (HT29 клетки се поддържат в среда, съдържаща 8% FBS топлинно инактивирана). Тези щамове бяха използвани в тестовете за свързване и неутрализиране на антитела AT12-015.
Генериране на антитела и получаване на Fab фрагменти.
Човешкото моноклонално антитяло AT12-015 срещу HPeV3 изолат 152037 е получено от човешка кръв 15. Накратко, CD27 + IgG + B клетки на човешката памет бяха култивирани, използвайки метода AIMSelect от здрави донори, които са се възстановили от изолирана 152037 инфекция с HPeV3, потвърдена 1 година по-рано. Единични клетки се субклонират от В клетъчни култури, в които супернатантът показва свързване с HPeV3 заразени клетки чрез имунофлуоресценция. РНК се изолира от моноклонални В клетки за възстановяване на тежките и леки верижни последователности на антитялото. Уникални последователности бяха използвани за генериране на рекомбинантен протеин в 293 Т клетки. IgG1 антителата бяха допълнително пречистени с помощта на колони HiTrap протеин А на инструмент ÄKTA (GE). Fab фрагментите от AT12-015 са получени с помощта на микропреп комплект Pierce Fab съгласно инструкциите на производителя, за да се постигне пълно усвояване на антитялото. Получените Fab фрагменти се смесват с HPeV3 капсиди в моларно съотношение 60 към 1 и се инкубират в продължение на 30 минути при 37 ° C в 1 х TNM, за да се позволи образуването на вирус-Fab комплекс, преди стъклообразуване.
Тест за неутрализация на HPeV
Неутрализацията на HPeV3 152037 изолат от AT12-015 беше тествана чрез заразяване на клетките на Vero с предварително инкубирани вируси (100 единици TCID50) с различни количества от антителата (0,03–7,5 µg ml -1) при 37 ° C за 1 h. Заразените клетки се наблюдават за появата на CPE на всеки 24 часа в продължение на 7 дни. На 7-ми ден РНК се екстрахира от супернатантата, като се използва комплект за изолиране на обща нуклеинова киселина с инструмента MagnaPure LC (Roche Diagnostics), използва се обратна транскрипция и cDNA (комплементарна ДНК) за оценка на броя на вирусното копие чрез PCR в реално време с помощта на Инструмент LC480 (Roche Diagnostics) 16. Способността на AT12-015 да неутрализира HPeV3 изолат 152037 също е тествана в BGM (биволско-зелен маймунски бъбрек), A549 (човешки аденокарцином на дебелото черво) и Caco2 (аденокарцином на човешкото дебело черво) клетъчни монослоеве. Освен това, кръстосана неутрализация от AT12-015 срещу HPeV1, HPeV2, HPeV4, HPeV5 и HPeV6 също е тествана в клетки Vero. Имунофлуоресценцията на заразени с HPeV1-6 клетки също беше проверена с помощта на AT12-015.
Криоелектронна микроскопия и обработка на изображения.
Пречистеният, инактивиран с формалдехид HPeV3 се витрифицира чрез нанасяне на 3 µl от пробата върху стелажи Quantifoil R3.5/1, изсушава се в продължение на 2 s при 92% относителна влажност и се потапя в течен етан с помощта на Leica EM GP. Решетките бяха изследвани на FEI Titan Krios трансмисионен електронен микроскоп, коригиран за Cs при 300 keV. Изображенията са записани на детектор Falcon II при условия на ниски дози с номинално увеличение 59 000 × с размер на пробата от 1,14 Å на пиксел. Седем кадъра на изображение бяха събрани в режим на преброяване с помощта на автоматизиран софтуер за събиране на единични частици FEI EPU. Първоначално филмовите рамки бяха подравнени един с друг с помощта на софтуера motioncorr 17 преди последващата обработка.
За асиметричната реконструкция ориентациите на най-добрите 41 845 частици бяха усъвършенствани за допълнителни 16 кръга, като се използва триизмерната опция за автоматично усъвършенстване в Relion, без да се налага симетрия. Окончателното усъвършенстване между два независими набора от данни даде резолюция от 10,36 Å въз основа на критерия FSC 0,143.
Аликвотни части от комплекси HPeV3-Fab AT12-015 бяха витрифицирани върху топлинно разтоварен ултрафин въглероден филм, съдържащ медни и въглеродни решетки (TED PELLA), използвайки самоизработена гилотина. Решетките бяха изследвани на FEI Tecnai F20 електронен микроскоп за пропускане при 200 keV с помощта на криостат Gatan 626. Изображенията бяха записани на филми Kodak SO-163 при условия на ниски дози с номинално увеличение 50 000 ×. Филмите бяха дигитализирани на Zeiss скенер (Photoscan) с размер на стъпка от 7 µm, което дава размер на пиксела от 1,4 Å на пиксел. Контрастната трансферна функция на всяка микрография се изчислява, както е описано по-горе. Частиците бяха събрани на микроснимки на интервали 4 ×, използвайки програмата RobEM 21 с размер на кутията 101 пиксела. Модел на случаен старт беше генериран от 150 частици, използвайки AUTO3DEM v 4.05.1 (реф. 21). Пълният набор от данни беше разделен на две и ориентациите и произхода бяха определени от независими реконструкции, генерирани от всеки набор. Крайната разделителна способност от 15 Å от 564 частици е постигната въз основа на критерия 0,143 FSC.
Изграждане на модела
Структурите на трите HPeV3 капсидни протеина бяха предсказани чрез сравнително моделиране на множество шаблони, използващи сървъра I-TASSER 25. Структурите на шаблона за VP0 включваха потребителски дефиниран шаблон, съдържащ кондензирана структура на VP4 човешки ентеровирус (PDB ID: 3vbf) N терминал за промиване на човешки ентеровирус VP2 (PDB ID: 3vbo) 26, както и болест на шапа. вирус (PDB ID: 1qqp) 27, полиовирус 1 (PDB ID: 1pov) 28, говежди ентеровирус (PDB ID: 1bev) 29, еховирус 1 (PDB ID: 1ev1) (справка 30) и хепатит A (PDB ID: 4qpg ) 31. Резултатът C за най-добрия модел беше –1.03.
За VP3 е използван празен предоставен от потребителя шаблон за човешки ентеровирус (PDB ID: 3vbo) 26, в допълнение към следните шаблони, избрани от програмата: coxsackievirus A9 (PDB ID: 1d4m) 32, echovirus 1 (PDB ID: 1d4m) PDB: 1ev1) (справка 30), човешки ентеровирус (PDB ID: 3vbf) 26, човешки ентеровирус (PDB ID: 3vbh) 26, човешки коксаки вирус A16 (PDB ID: 4jgy) 7. Резултатът C за най-добрия модел беше –0,61.
За VP1 празен човешки ентеровирус (PDB ID: 3vbo) 26 беше предоставен като външен шаблон в допълнение към следните шаблони, избрани от програмата: човешки ентеровирус 71 (PDB ID: 3vbh) 26, човешки ентеровирус 71 (PDB ID: 3zfe) 33, човешки коксаки вирус A16 (PDB ID: 4jgy) 7, човешки ентеровирус 71 (PDB ID: 4cdq) 34, coxsackievirus A 9 (PDB ID: 1d4m) 32, човешки риновирус 14 (PDB ID: 1ncq) 35. Резултатът C за най-добрия модел беше –0,75.
Моделите на хомологията бяха твърдо монтирани на картата HPeV3, използвайки функцията „годни за картографиране“ в UCSF-Chimera 36. Използвайки функцията „зониране“ в UCSF-Chimera 36, капсидната карта HPeV3 беше разделена на асиметрична единица с радиус 6 Å, използвайки модела VP0-VP3-VP1 с твърдо прилягане. Моделът VP0-VP3-VP1 беше гъвкаво монтиран в асиметричния блок, използвайки сравнението на резултатите от две различни програми за гъвкаво приспособяване iMODfit 37 и FlexEM 38, за да се постигне консенсусно прилягане 39. И двете гъвкави програми за настройка бяха използвани с настройките по подразбиране. След това моделите бяха прецизирани ръчно в Coot 40 чрез отрязване на шаблоните, където плътността не беше очевидна, подобряване на прилепването на гръбнака и регулиране на тежки странични вериги от фенилаланин, тирозин, триптофан и аргинин остатъци, където са видими. Моделите бяха допълнително усъвършенствани чрез усъвършенстване на реалното пространство в Phenix 41. Всички визуализации бяха извършени в UCSF-Химера 36 .
Едно копие на РНК структурираната област беше извлечено с помощта на „Bulk Eraser“ в UCSF-Chimera 36. Извлеченият обем може да съдържа около 30 нуклеотида. 30 нуклеотиден регион (7181-7210) в 3 'нетранслирания край на изолирания HPeV3 геном 152037 (GenBank ID: GQ183026) беше сгънат на RNAfold уеб сървър 42 и сгъването беше моделирано в RNAComposer 43. Този модел на РНК е монтиран на извлечения обем с помощта на опцията „Fit to Map“ в UCSF-Chimera 36 .
Отпечатъкът AT12-015 беше оценен чрез наслагване на атомния модел HPeV3 при реконструкцията на комплекса HPeV3-Fab AT12-015 и генериране на пътна карта за атомния модел с легендарния отпечатък като контурни линии в RIVEM 44. Всички аминокиселинни остатъци, които бяха изложени на повърхността в рамките на този отпечатък, бяха взети като епитопи за Fab.
Допълнителна информация
Кодове за достъп: Картите на плътността на икосаедрично-симетричните HPeV3, асиметричните HPeV3 и HPeV3-Fab AT12-015 са депозирани в Банката данни за електронна микроскопия под кодовете за достъп EMD-3137, EMD-3322 и EMD-3138, съответно. Моделите, адаптирани за HPeV3, са депозирани в банката с данни за протеини в Европа под PDB ID: 5APM. Всички сурови данни, събрани за реконструкцията на HPeV3, са достъпни чрез пилотен файл с електронна микроскопия с код за достъп EMPIAR-10033.
Как да цитирам тази статия: Shakeel, S. et al. Множество взаимодействия, стабилизиращи капсидите, разкрити в структура с висока разделителна способност на възникващ пикорнавирус, причиняващ неонатален сепсис. Нат. Комун. 7: 11387 doi: 10.1038/ncomms11387 (2016).
- Папая, плод с множество ползи за здравето, който трябва да включите в ежедневната си диета -
- Многобройните и полезни свойства на спермата Placeres S Moda EL PAÍS
- Какъв е идеалният прием на калории за човек с височина 5 фута и 6 инча с тегло 95 кг?
- Жените, които искат да отслабнат, трябва да ядат диета с високо съдържание на млечни протеини -
- Девет плода с множество ползи за кожата